版权所有 © 2023 范德耗材 版权所有 津ICP备2023001149号-1
单细胞测序技术是揭示肿瘤遗传和功能异质性的重要工具,单细胞测序技术为癌症的克隆进化、微环境、耐药性和转移进展提供了独特的见解。微流体是许多SCS技术和工作流程的关键组成部分,在吞吐量、经济性和自动化方面具有优势。在这里,我们了解一下用于单细胞组学分析的微流体结构和测序技术的现状。
SCS平台和工作流程的开发与基于微流体的技术齐头并进。微流体技术特别有助于解决SCS低吞吐量的挑战,但也提供了集成和自动化的潜力,以提高SCS的性能。
接下来,我们重点讲解一下基于微流体的SCS在癌症中的应用。
基因组
单细胞DNA测序(scDNA-seq)在不同水平上深入了解基因组的细胞间变异和异质性,包括单核苷酸变异(SNV)和拷贝数畸变(CNA)。scDNA-seq可以说是揭示肿瘤进化、治疗或转移的克隆亚结构和谱系的最直接方法。
然而,由于细胞中只有很少的DNA分子拷贝,因此对基因组进行测序是一项挑战。因此,全基因组扩增(WGA)是scDNA-seq的关键部分。目前使用的最常见的WGA方法是多重置换扩增(MDA)和变性寡核苷酸引物聚合酶链式反应(DOP-PCR)。较新的方法包括多重退火和基于环的扩增循环(MALBACs)和通过转座子插入的线性扩增(LIANTI)。WGA通常面临一些挑战,例如测序深度的变异性、等位基因脱落(两个等位基因不会同时扩增)、扩增偏差等。此外,基因的结构变异(SV)包括缺失、重复、插入和易位,这可能很难用WGA方法识别。
已经提出了几种技术来解决这些问题。未扩增的Tn5标记的scDNA-seq方法,如基于微流体的DLP和DLP+(图2A),可以避免基于扩增的错误。Ruan等人开发了一种DMF芯片,该芯片能够分离具有流体动力学陷阱蝴蝶结构的单细胞(图2B)。在该设计中,通过致动液滴将细胞与裂解物和MDA试剂混合来扩增细胞。单细胞纳升体积MDA处理的整个步骤是以集成和非接触的方式实现的。这可以减少指数扩增中的扩增偏差和误差,并提供对CNV和SNV的极好检测。
图1。基于微流体的SCS在癌症精准医疗中的应用示意图

图2:基于微流体的SCS技术示意图
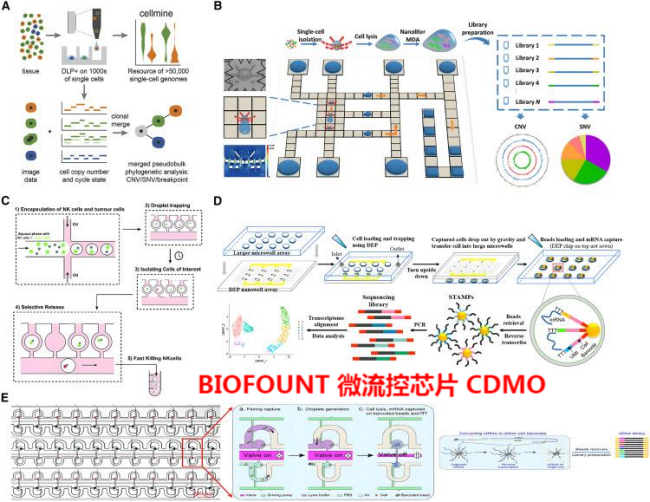
(A) 用于DNA测序的微孔微流体和基于Tn5转座酶的DLP+方法的示意图。数千个细胞中的高DLP+吞吐量和大的基因组覆盖率。
(B) 用于单细胞全基因测序的DMF平台的图示。
(C) 用于自下而上分析感兴趣肿瘤细胞选择的微流体平台工作流程示意图。
(D) 基于DEP和纳米阱的dTNT-seq的工作流程图。
(E) 成对序列芯片的示意图。该系统包括流体动力学捕获,通过阀门和泵控制流体。
scDNA-seq的一个关键贡献是揭示靶向治疗后肿瘤细胞耐药性的克隆进化。scDNA-seq与微流体一起提供了实现癌症动态治疗的直接临床相关性的可能性。例如,McMahon等人通过利用商业两步液滴微流体平台(Tapestri)对接受吉地替尼临床治疗的FLT3突变急性髓系白血病(AML)患者进行了高通量scDNA-seq。该结果确定了吉地替尼治疗对RAS/MAPK通路信号的激活,这是一种具有临床意义的耐药性机制,并表明治疗AML需要一种增强抗白血病细胞毒性的组合方法。

转录组
单细胞RNA测序(scRNA-seq)是肿瘤研究和治疗中最流行的测序方式。在肿瘤学治疗研究中,scRNA-seq通过分析决定肿瘤内细胞表达和功能异质性的因素提供了一个有价值的视角。
在scRNA-seq中,首先需要将RNA转化为cDNA。根据逆转录和扩增的不同方法,scRNA-seq大致可分为三组。聚(A)缀合的聚合酶链式反应是一种选择,但由于测序灵敏度降低和低丰度转录的损失,它不常被采用。体外转录(IVT)是一种线性扩增过程,涉及5′端含有T7启动子的复杂引物。该方法适用于通过线性扩增和测序(CEL-seq)扩增细胞表达的步骤。最后,Moloney小鼠白血病病毒(MMLV)逆转录RNA可以通过包括完整的5′端来合成全长cDNA,从而降低转录的3′偏误。基于MMLV的逆转录已用于Smart Seq、Smart-Seq2和单细胞标记逆转录测序(STRT-Seq)工作流程。为了克服偏差问题,在逆转录步骤中加入了一种独特的分子识别器(UMI),以对细胞中的每个信使核糖核酸分子进行条形码标记,增强了scRNA-seq的定量性质,并提高了读数的准确性。
在scRNA-seq中,两种最常见的方法是基于液滴的scRNA-seq和基于微孔的scRNA-seq。基于微孔的scRNA-seq的准确性和灵敏度可能更高,但商业化的成本比基于液滴方法更高。InDrop、Drop-seq、和10×Genomics Chromium是更常见的基于液滴的技术,因为它们成本低、操作简单、吞吐量高。所有三个平台都涉及使用条形码珠引物来区分单个细胞和用于偏差校正的UMI。除了更标准的基于液滴的方法外,许多小组一直在努力在其scRNA-seq工作流程中利用替代的微流体设计原理。Li等人通过使用未完全被油包裹的细胞和虚拟液滴的流体动力学捕获进行选择和下游信使核糖核酸测序,实现了对具有不同MMP9分泌效率的肿瘤细胞的转录基因分析。该微流体平台具有648个微腔,单细胞捕获效率高达90%,以及检索感兴趣细胞的能力,为临床诊断中评估肿瘤恶性提供了新的可能性。此外,Bai等人展示了一种集成的DEP捕获纳米阱转移(dTNT)技术,以打破泊松极限,实现单细胞捕获率91.84%、转移效率82%和>99%珠粒负载的scRNA-seq(图2D)。

基于瓣膜的scRNA-seq由于其在有效的细胞捕获和选择感兴趣的细胞进行分析方面的潜力而被越来越多地采用。张教授等人利用流体动力学捕获和基于瓣膜的设计创建了一个高度平行的scRNA-seq平台,称为“成对的seq”。单个细胞和单个条形码珠都被捕获在成对的腔室中,然后瓣膜和泵的组合系统能够将它们积极混合,以允许细胞裂解和捕获信使核糖核酸。该系统提供高达95%的细胞利用效率和高的信使核糖核酸检测准确性(图2E)。Agnihotri等人的另一种基于阀门的微流体芯片通过将自然杀伤(NK)细胞和肿瘤细胞共同封装在液滴中,为“自下而上”的SCS设计了一个三层微流体系统。它通过使用微阀选择性释放感兴趣的细胞用于下游测序分析(图2C)。在这项工作中,免疫细胞与肿瘤细胞的视觉观察,结合选择感兴趣的细胞进行随后的scRNA-seq,为免疫疗法提供了新的见解。
在接下来的文章中我们继续讲解一下基于微流体的SCS在癌症中的应用。